审稿人意见里面提到的一篇非常重要的文章,主要写了什么呢,一起来看看吧!
作者声称他们的pepflow是第一个用了flow matching的多模态深度生成模型,他们的目的是:在已知目标蛋白质受体的条件下,设计出相应的肽(和我们的非常类似orz
他们的动机是现有的生成模型比如Diffusion仅关注骨架设计,忽略了侧链动态和全原子结构,强调需要同时建模序列与结构、结合位点条件化生成,以及多模态联合分布的必要性。
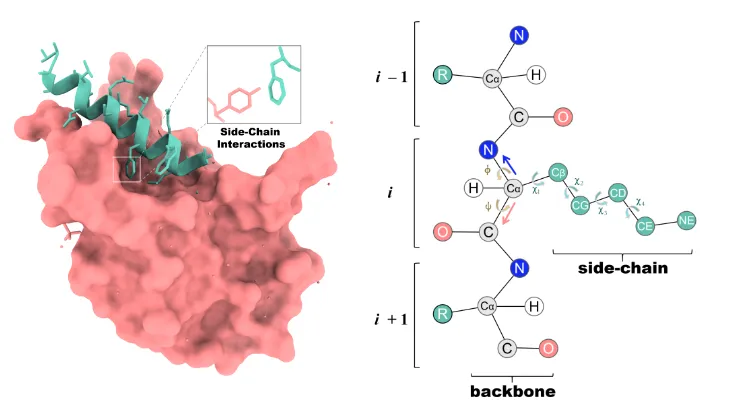
引言部分的图片用于引出问题,左边展示的就是肽和目标蛋白的结合示意图,突出关键残基的骨架方向和侧链相互作用。右图呢说明了每个蛋白质残基包含了骨架原子和侧链原子,意思就是说肽和蛋白质相互结合的关键因素就是这俩,那在这个工作里面就要把他们考虑进去。
方法部分定义将肽的每个残基表示为刚性框架(位置和方向)、侧链角度和离散类型,并为每种模态构建分析流
肽的数学表示:残基类型()、骨架刚体变换()、侧链扭转角(),并建模条件分布。
他这里的多模态是这样定义的,在四个维度进行建模:位置、方向、角度、类型
位置(Position, Euclidean CFM):使用欧几里得流匹配(线性插值)。
方向(Orientation, Spherical CFM):在SO(3)流形上通过测地线插值定义流。
角度(Angles, Toric CFM):在环面()上处理周期性角度。
类型(Type, Simplex CFM):将离散残基类型映射到概率单纯形(),通过logit空间进行流匹配。
联合目标函数:加权多模态损失(位置、方向、角度、类型)。
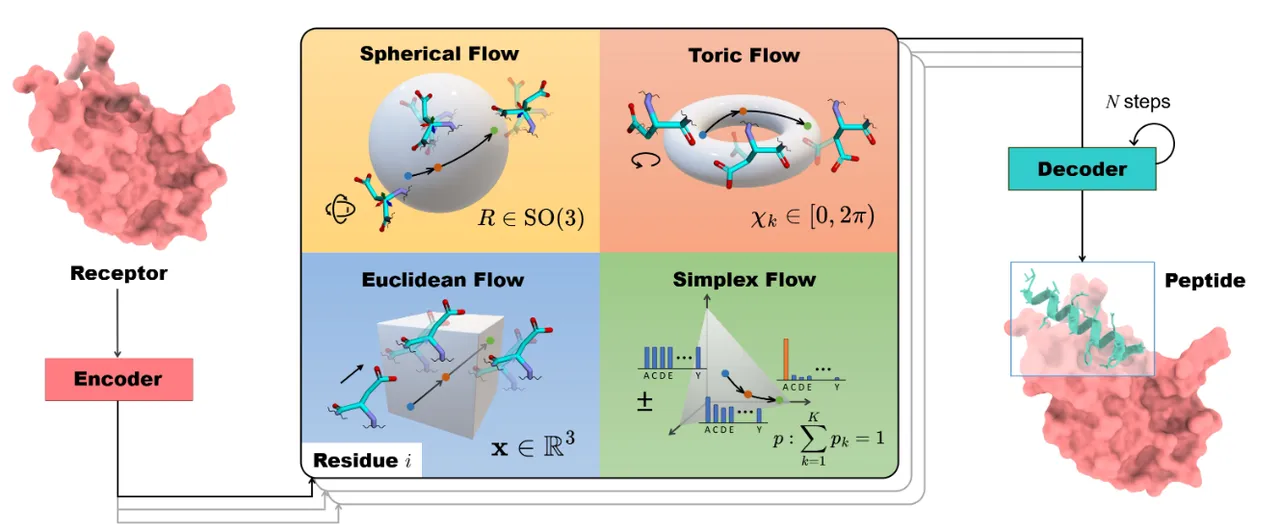
他的架构其实很简单,就是encoder和decoder,encoder是用于提取目标蛋白质的几何特征也就是编码为embedding,decoder基于Invariant Point Attention对各个模态的flow进行预测,通过欧拉方法迭代生成肽结构
实验分为三大部分:序列结构协同设计、固定骨架序列设计、侧链包装
